Table des matières
Utilisation de l'IBM Blue Gene/P de l'IDRIS : Le projet CSCV
Contrôle Spatialisé d'un Vecteur de la maladie de Chagas dans un village réel (CSCV)
Corentin Barbu 1a, Eric Dumonteil 2bSamira El-Yacoubi a et Sébastien Gourbière3a a. Laboratoire de Mathématiques, Physique et Systémes (EA 4217), Université de Perpignan Via Domitia, 52 Avenue Paul Alduy, 66860 Perpignan Cedex, France Tél : 04.68.66.17.64. b. Centre de recherche régional « Dr. Hideyo Noguchi », Université autonome du Yucatan, Mexique. 1. responsable technique corentin.barbu@univ-perp.fr 2. collaborateur au mexique pour l'obtention des données. 3. responsable de projet gourbier@univ-perp.fr
Résumé du projet
Avec 10 à 11 millions de personnes infectées (Moncayo & Yanine 2006), la maladie de Chagas est la maladie vectorielle la plus importante d'Amérique latine. En l'absence de vaccins et de traitements, l'organisation Mondiale de la Santé (OMS) recommande de lutter contre les insectes transmettant le parasite par le biais de pulvérisations d'insecticides. Cette stratégie a permis une réduction importante du fardeau associé à la maladie ces dernières années (Schofield et al. 2006).
Cependant, dans plusieurs régions d'Amérique latine, l'immigration régulière d'insectes depuis les zones non traitables (telles que le milieu forestier) limite considérablement l'efficacité de ces programmes (Gürtler et al. 2008). C'est dans ce contexte que s'insère ce projet qui implique un groupe de parasitologistes mexicains et notre équipe de biomathématiciens, avec l'objectif commun de contrôler les populations d'insectes vecteurs de la maladie dans la péninsule du Yucatan (Mexique). Un modèle stochastique spatialement explicite a été développé qui, adossé à des jeux de données exceptionnels et alimenté par les moyens mis à disposition par l'IDRIS en 2008, nous a permis de caractériser la dispersion des vecteurs dans un des villages d'étude.
Objet de la recherche, problématique scientifique
La collaboration menée depuis plusieurs années par les membres de ce projet a permis de démontrer dans plusieurs villages de l'état du Yucatan la présence temporaire de punaises vectrices de la maladie causée par l'immigration saisonnière d'insectes dans les maisons (Gourbière et al. 2008). Alors que cette immigration est très bien caractérisée dans le temps (elle a lieu entre avril et juin) sa dimension spatiale est jusqu'à maintenant très peu connue. Par exemple, la provenance des insectes et leurs capacités migratoires sur le terrain sont encore sujet à discussion. Or l'absence de connaissances précises sur la distribution spatiale des vecteurs et leurs capacités de dispersion empêche d'élaborer des politiques de contrôle efficaces et soutenables dans le temps. Dans l'objectif de saisir la dynamique spatiale de l'immigration, nos partenaires mexicains assistés par la population locale ont effectués des comptages maison par maison et tous les 15 jours pendant une année sur l'ensemble du village de Teya dans l'état du Yucatan au Mexique. Un modèle spatialisé individu centré a alors été développé afin de tester sur ces données la vraisemblance (LikelyHood) d'hypothèses qualitatives telles que, par exemple, la possibilité que les punaises viennent uniquement des jardins du village et d'estimer quantitativement des paramètres essentiels tels que, par exemple, la vitesse moyenne de déplacement des punaises.
Caractéristiques du code et de l?implémentation sur la Blue Gene/P
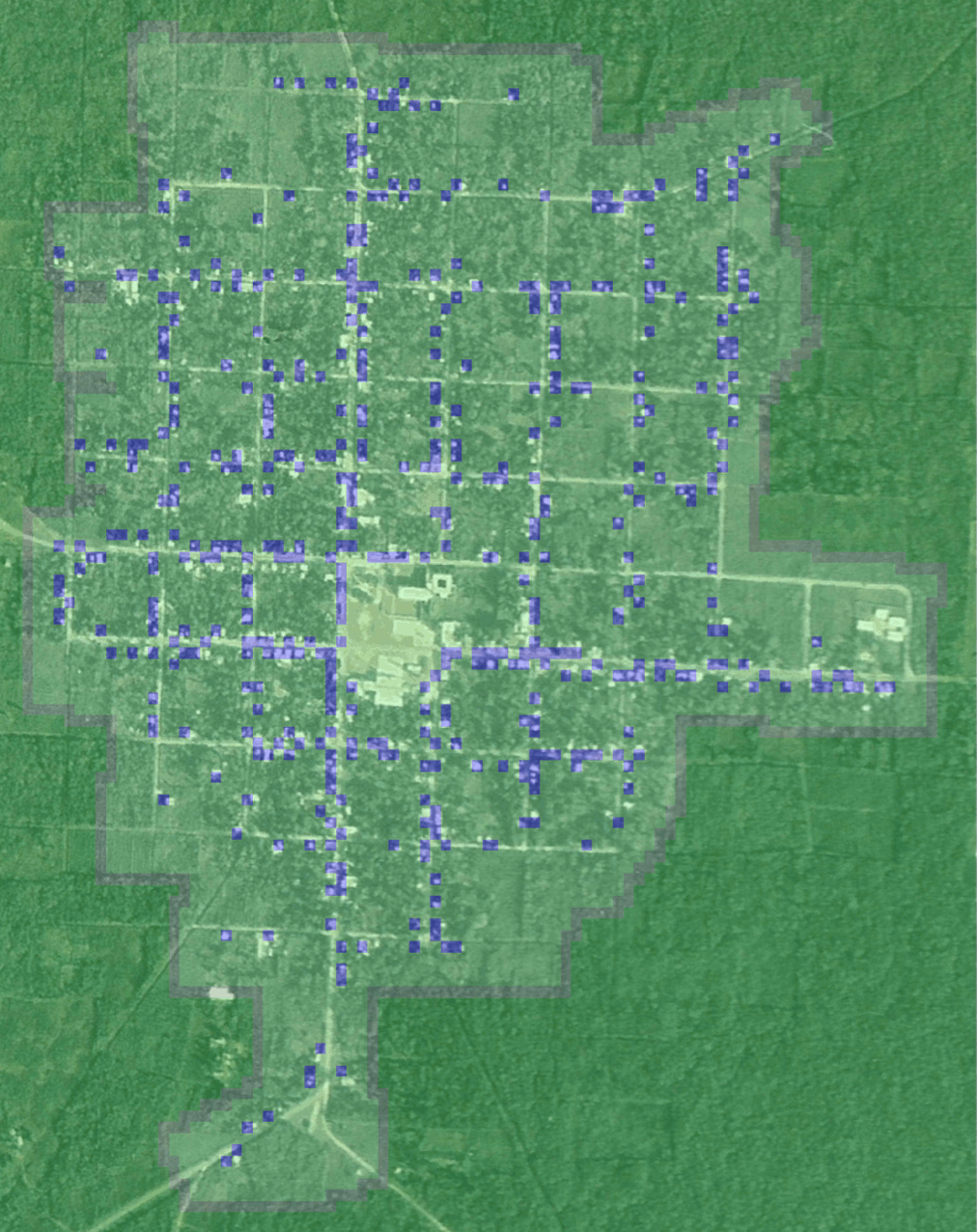
Un pointage GPS des maisons sur une photo aérienne a permis de disposer d'une représentation numérique fidèle des différents types d'habitat rencontrés par les vecteurs à l'intérieur du village (fig.1). Sur cet espace les déplacement des punaises ont été modélisés individu par individu. Etant donné le caractère stochastique des processus sous-jacents, un grand nombre de répliques était nécessaire pour obtenir la succession des cartes moyennes de déplacement au cours de l'année, comparable avec les données. De plus, le nombre élevé de paramètres (9), impliquait un nombre important de calculs pour trouver les paramètres optimisant la correspondance entre données et sorties du modèle. Pour effectuer ce très grand nombre de simulations, une structure maitre/esclave à deux niveaux a été implémentée en C et MPI. L'espace mémoire nécessaire pour chaque simulation étant restreint, notre implémentation a pu exploiter pleinement les capacités des 1024 coeurs utilisés couramment (mode VN).
Figure 1 : Superposition de la carte numérique réalisée et de la photo aérienne du village de Teya, Yucatan, Mexique.
Description des résultats obtenus
Une bonne correspondance entre le modèle optimisé et la dynamique temporelle d'infestation du village a été obtenue à l'échelle de l'ensemble du village et des grandes zones géographiques du village (résultats non présentés ici). Les données et des sorties types sont disponibles sous forme d'animations. L'utilisation du critère d'information d'Akaike permet de rejeter les modèles faisant l'hypothèse d'une source unique des punaises (forêt ou intérieur du village, voir tableau 1), renseignements précieux pour l'élaboration ultérieure de stratégies de contrôle efficaces. En revanche, la non prise en compte d'un facteur d'attraction par les maisons abaisse la vraisemblance mais pas suffisamment pour pouvoir affirmer avec certitude que les punaises sont attirées par les maisons.
| Hypothèse | AlCc | Poids du modèle |
| générale | 1442,18 | 0,83 |
| sans forêt | 1461,46 | 0 |
| sans jardin | 1483,15 | 0 |
| sans attraction par les maisons | 1445,36 | 0,17 |
Tableau 1: Evaluation des performances des différentes hypothèses qualitatives La première colonne donne, sous un énoncé simplifié, les hypothèses correspondant à chaque modèle. La deuxième colonne donne les critères d'information d'Akaike (AIC). La troisième colonne donne le poids du modèle, qui peut être vu comme la probabilité que le modèle considéré soit le meilleur parmi ceux envisagés.
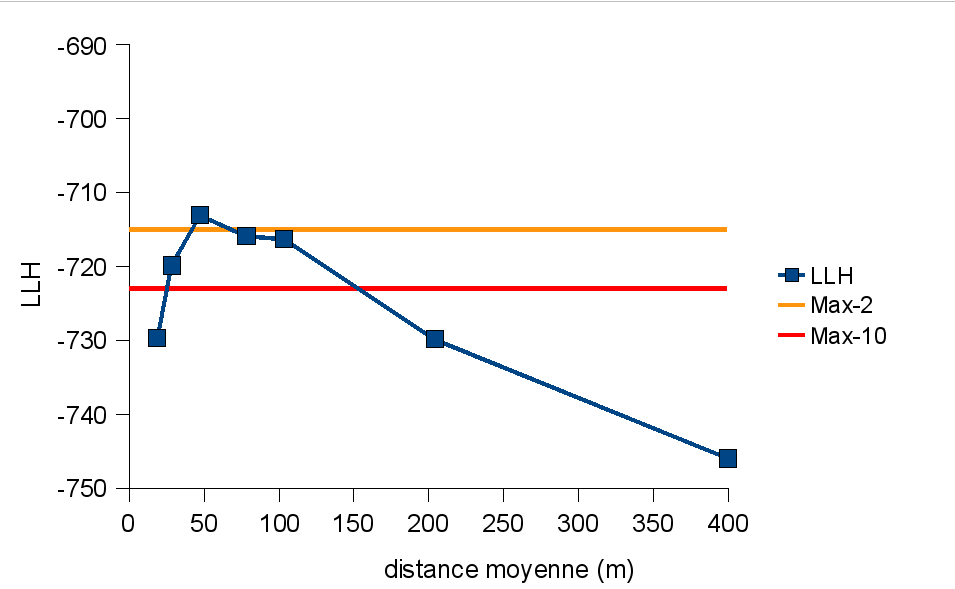
Par ailleurs, les valeurs de paramètres nécessaires pour atteindre un bon ajustement entre les sorties du modèle et les données correspondent à des distances moyennes de déplacement assez faibles par rapport aux capacités potentielles des punaises. Une série d'ajustement modèle/donnée pour des distances moyennes de déplacement fixées a donc été menée afin de visualiser l'influence de la distance moyenne sur la qualité de l'ajustement. Ce travail permet de voir que les données contraignent fortement la distance moyenne autour de 50 m et qu'une distance moyenne de déplacement de 200 m ou plus n'est pas envisageable (fig. 2), alors que les observations de terrain le laissaient penser. Là encore, cette information est précieuse pour envisager un contrôle de ces migrations, ce qui est la prochaine étape de notre travail.
Figure 2: Valeurs des LLH obtenus pour des distances moyennes imposées Les lignes oranges et rouges marquent respectivement les seuils de ?différence probable? et de ?différence significative?. LLH: Log LikeliHood, les faibles valeurs absolue correspondent à la plus grande vraisemblance. La distance moyenne est la distance moyenne en mètres parcourue par une punaise en 15 jours.
Références et publications associées
Gourbière, S.; Dumonteil, E.; Rabinovich, J. E.; Minkoue, R. & Menu, F. (2008). Demographic and dispersal constraints for domestic infestation by non-domicilated chagas disease vectors in the Yucatan peninsula, Mexico, American Journal of Tropical Medicine and Hygiene 78 : 133-139.
Gurtler, R. E.; Diotaiuti, L. & Kitron, U. (2008). Commentary: Chagas disease: 100 years since discovery and lessons for the future, Int. J. Epidemiol. 37 : 698-701.
Moncayo, A. & Yanine, M. I. O. (2006). An update on Chagas disease (human American trypanosomiasis)., Annals of Tropical Medicine and Parasitology 100 : 663-677.
Schofield, C. J.; Jannin, J. & Salvatella, R. (2006). The future of Chagas disease control., Trends in Parasitology 22 : 583-588.