Table des matières
Utilisation de l'IBM Blue Gene/P de l'IDRIS : Le projet Nico_bacter
Simulation d'un analogue bactérien du récepteur nicotinique de l'acétylcholine à pH 4.6 et vérification de la stabilité de l'état ouvert en l'absence de détergents à l'intérieur du canal (Nico_bacter)
Responsable : Marc Delarue, Unité de Dynamique Structurale des Macromolécules, Institut Pasteur, Paris.
Collaborateurs : Hugues Nury, Unité de Dynamique Structurale des Macromolécules, Institut Pasteur, Paris. Marc Baaden, Laboratoire de Biochimie Théorique, IBPC, Paris.
Résumé du projet
Le laboratoire a récemment résolu la structure tridimensionnelle à 2.9 A par diffraction aux Rayons X d'un analogue bactérien du récepteur nicotinique de l'acétylcholine en présence de détergent. La structure a révélé un pentamère dans une conformation ouverte, par opposition à la structure d'un autre récepteur bactérien homologue (Hilf et Dutzler, 2008), résolu à 3.3 A, et qui se trouve dans une conformation fermée. En principe, le canal est dans une conformation telle qu'il permettrait le passage de cations. Cependant, la structure a révélé également la présence de 6 molécules de détergent (une par monomère, plus une centrale, sur l'axe 5) à l'intérieur du canal, bouchant effectivement celui-ci.
Nous avons voulu tester si la conformation ouverte reste stable lors d'une simulation tout atomes (~250,000 atomes) en présence d'eau et de lipides, quand on enlève les détergents fixés à l'intérieur du canal.
Objet de la recherche, problématique scientifique
Dans un premier temps, l'objet de ce projet est de compléter l'étude cristallographique par une étude en dynamique moléculaire du récepteur bactérien, avec et sans molécules de détergents dans le canal, afin de vérifier la stabilité de l'édifice dans des conditions proches de la réalite biologique.
Caractéristiques du code et de l?implémentation sur la Blue Gene/P
Le code utilisé est NAMD developpé par l'équipe de Klaus Schulten aux Etats Unis; il a déjà été optimisé pour une machine telle que Blue Gene d'IBM. En effet, il est spécialement conçu pour simuler de grands complexes biomoléculaires sur des machines parallèles comprenant jusqu'à plusieurs centaines de processeurs. Le code est disponible gratuitement sur http://www.ks.uiuc.edu/Research/namd/.
Description des résultats obtenus
Un modèle atomique complet incluant les atomes d'hydrogène a été construit à partir de la structure cristallographique du canal ionique d'interêt. Les calculs de pKa à pH 4,6 ont été réalisés avec le logiciel Yasara afin de déterminer les états de protonation les plus probables des résidus titrables. Le modèle de la proteine a été inséré dans une bicouche de POPC et hydrate (307 molécules de lipides et 43992 molécules d'eau). L'ajout d'ions sodium et chlorure a permis de rendre le système neutre. La construction du modèle a été réalisée avec le programme VMD. Les simulations ont ensuite été menées avec le logiciel NAMD en utilisant le champ de force CHARMM2748. Deux étapes d'équilibration du système, avec la proteine bloquée puis en relâchant progressivement cette contrainte, ont précédé la phase de production de 20 ns de simulation. Un film est disponible sur http://www.shaman.ibpc.fr/glic/page1/page1.html.
Le résultat principal obtenu est que la conformation ouverte est stable même en l'absence de molécules de détergents à l'intérieur du canal, démontrant ainsi que cet artefact de cristallisation n'empêche pas de tirer des conclusions sur les changements conformationnels observés dans notre structure cristalline (ouverte) par rapport à celle qui a déjà été publiée par le groupe de Dutzler un peu plus tôt cette année (fermée).
Ce travail fait partie d'un tout qui a donné lieu à une publication le 05/11/08 dans Nature.
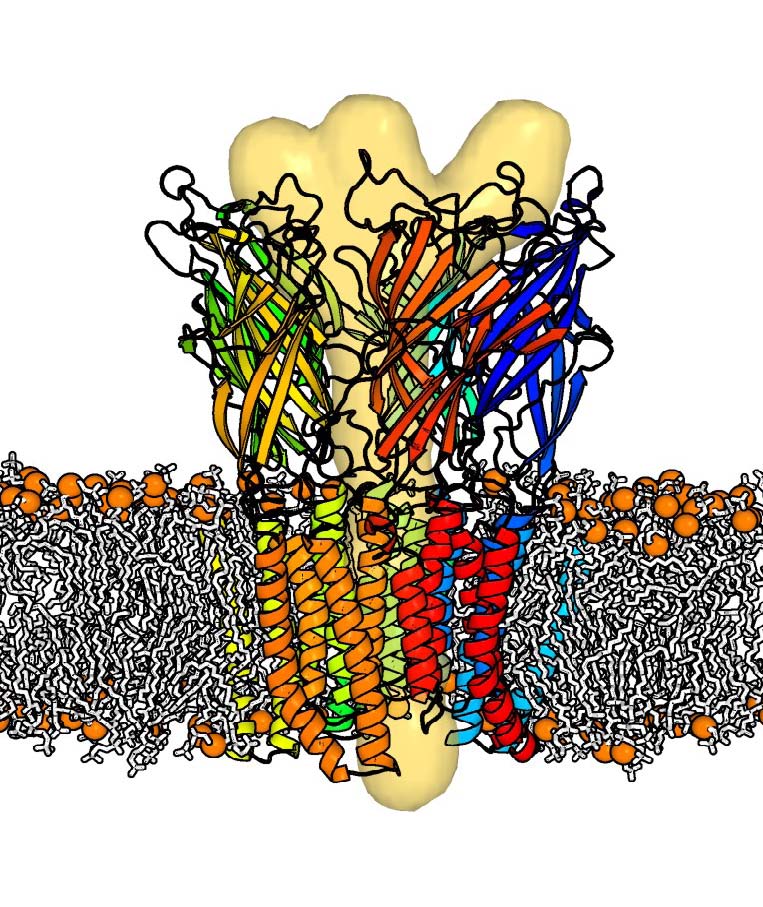
Légende de l?image
Représentation du récepteur pentamérique inséré dans une bicouche lipidique. L'enveloppe de la cavité d'entrée et du canal est également représentée.
Références et publication associée
Xray structure of a pentameric ligandgated ion channel in an apparently open conformation N. Bocquet, H. Nury, M. Baaden, JP Changeux, M. Delarue and P.J. Corringer. Nature, 2008 in the press. L'article sera disponible en ligne le 05/11/08.